ยีนที่เกี่ยวข้องกับการสร้างสีแอนโทไซยานินของดอกกล้วยไม้สกุลหวาย และสกุลแวนด้า |
ประเทศไทยเป็นแหล่งผลิตกล้วยไม้เมืองร้อนที่สำคัญของโลก มีการปลูกเลี้ยงคิดเป็นพื้นที่ประมาณ 17,500 ไร่ และทำรายได้ประมาณปีละ 2,000 ล้านบาท อุตสาหกรรมกล้วยไม้ไทยกำลังเผชิญปัญหาการขาดแคลนกล้วยไม้พันธุ์ใหม่ๆ ที่มีสีดอกแปลกตา แนวทางแก้ไขก็คือการแสวงหาพันธุ์ใหม่ เทคนิคทางพันธุวิศวกรรมจึงเป็นทางเลือกในการปรับปรุงพันธุ์ของดอกกล้วยไม้ โดยเฉพาะสีของดอกกล้วยไม้ให้มีความหลากหลายมากขึ้นกว่าพันธุ์เดิม เนื่องจากประเทศไทยยังขาดข้อมูลยีนสร้างสีดอกของกล้วยไม้ งานวิจัยในครั้งนี้จึงมุ่งเน้นในการโคลนยีนที่ควบคุมการสร้างสีดอกในกลุ่มแอนโทไซยานินของดอกกล้วยไม้สกุลหวาย และลูกผสมกล้วยไม้สกุลแวนด้า ภาพที่ 1 กระบวนการสังเคราะห์สารแอนโทไซยานิน (Holton and Cornish, 1995) และ ชนิดของยีนที่โคลนในกล้วยไม้ คือ 1. CHS, 2. F3’H, 3.DFR, 4. ANS/FLS
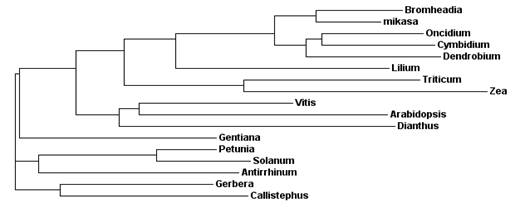
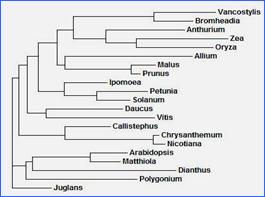
ภาพที่ 2 Phylogenetic tree แสดงระดับความสัมพันธ์ระหว่างโปรตีนของยีน dfr ในดอกกล้วยไม้แวนด้ากับ นอกจากนี้ยังโคลนยีนในช่วงท้ายของกระบวนการสร้างสีดอก คือ F3H และ F3¢5¢H จากกล้วยไม้หวายแอสโคเซนด้า และ อัญชัน (วาสนา, 2547) โดย เอไซม์ Flavonoid 3¢, 5¢-hydroxylase เป็นเอนไซม์ที่สำคัญในการสร้างรงควัตถุที่ให้ดอกไม้มีสีน้ำเงินหรือสีม่วง การโคลนยีน f3¢5¢h จากดอกอัญชันและดอกกล้วยไม้ไพน์ริเวอร์ เมื่อนำลำดับกรดอะมิโนของยีน f3¢5¢h จากดอกอัญชันและดอกกล้วยไม้ไพน์ริเวอร์มาเปรียบเทียบกันพบว่ามีความคล้ายคลึงกันคิดเป็นร้อยละ 90 และเมื่อนำลำดับกรดอะมิโนจากพืชทั้งสองชนิดไปเปรียบเทียบกับพืชชนิดอื่น พบว่ามีความใกล้เคียงกับเอนไซม์ F3¢H จากพิทูเนีย คิดเป็นร้อยละ 66 นอกจากนี้ยังสามารถโคลนยีน f3h จากดอกกล้วยไม้ไพน์ริเวอร์ เปรียบเทียบกับพืชอื่นพบว่ามีความใกล้เคียงกับเอนไซม์ F3H จากดอกกล้วยไม้ Bromheadia finlaysoniana คิดเป็นร้อยละ 94
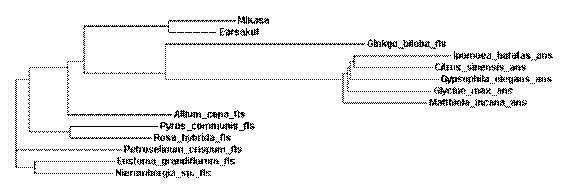
ภาพที่ 3 Phylogenetic tree แสดงระดับความสัมพันธ์ระหว่างโปรตีนของยีน f3h (ซ้ายมือ) River และ f3¢5¢h (ขวามือ) ในดอกกล้วยไม้ ของ Vancosylis Pine กับ โปรตีนของในพืชชนิดอื่น งานวิจัยครั้งนี้เน้นการโคลนยีน anthocyanidin synthase (ans) ที่เกี่ยวข้องกับขั้นตอนทางเคมีสุดท้ายของกระบวนการสร้างสารแอนโทไซยานินในดอกไม้ การรายงานครั้งนี้ ยีนใหม่ flavonal synthase (fls) ที่คล้ายกับยีน ans ได้โคลนจากดอกสีม่วงแดงของกล้วยไม้สกุลหวายเอียสกุล และดอกสีม่วงน้ำเงินของดอกสกุลแวนด้าลูกผสมมิกกาซา สายโพลีเปปไทด์ของเอนไซม์ FLS ที่ถอดรหัสของยีนโคลนจากกล้วยไม้ทั้งสองชนิดมีองค์ประกอบของกรดอะมิโน ความคล้ายคลึงกับโปรตีน FLS ร้อยละ 74-53 และ โปรตีน ANS ร้อยละ 50-46 ยีนที่โคลนได้นี้มีแนวโน้มที่เป็นเอนไซม์ฟลาโวนอยด์ที่เป็นกุญแจสำคัญต่อการเกษตรเพื่อการปรับปรุงสีดอกใหม่ของกล้วยไม้ในการศึกษาต่อไป อย่างไรก็ตามความสัมพันธ์ของโปรตีนที่ใช้ด้วยองค์ประกอบของโปรตีนเป็นหลักของศึกษาสามารถจัดจำแนกโปรตีน FLS และ ANS ออกเป็นสองกลุ่มเห็นได้ชัดเจน โดยมี FLS ของกล้วยไม้อยู่ระหว่างกลางดังแสดงให้เห็นโดย phylogenetic tree (ภาพที่ 4 ) เอนไซม์ FLS และ ANS มีความสำคัญต่อขบวนการสร้างสารแอนโทซัยยานิน เนื่องจากเอนไซม์ทั้งสองทำหน้าที่เปลี่ยนสาร dihydrokaempferol ซึ่งเป็นสารที่ไม่มีสีไปเป็นสีต่างๆได้ (Heller, 1985) แต่เอนไซม์ทั้งสองชนิดมีซับเตรตที่แตกต่างกัน โดย FLS มีซับเตรตเป็นสาร Dihydroquercetin และ Dihydromyricetin ส่วน ANS มีซับเตรตเป็นสาร Leucodelphinidin และ Leucocyanidin องค์ประกอบและลำดับของกรดอะมิโนของสายโพลิเปปไทด์ของเอนไซม์ทั้งสองชนิดมีที่คล้ายกันมาก มีความแตกต่างของโปรตีนในบางตำแหน่งเท่านั้น (Figure 3) โครงสร้างของโปรตีนที่แสดงความแตกต่างในแต่ละตำแหน่งถือเป็นประเด็นสำคัญที่ต้องศึกษาต่อไปในอนาคต โดยเฉพาะการศึกษาองค์ประกอบของยีนที่แยกจากล้วยไม้ซึ่งองค์ประกอบความคล้ายคลึงทั้ง FLS และ ANS
ภาพที่ 4 Phylogenetic tree แสดงระดับความสัมพันธ์ระหว่างโปรตีนของยีน ans ในดอกกล้วยไม้หวายเอียสกุล และ มิกาซา กัยโปรตีนของยีน ans และ fls ในพืชชนิดอื่น
|
| คณะผู้วิจัย : พัฒนา ศรีฟ้า ฮุนเนอร์ วาสนา เจียมตัว นครินทร์ ทองแถม ณ อยุธยา อุดมพร เพ็ชรไทย และ สุเจตน์ เจียมกิจรุ่ง หน่วยงาน : โครงการสหวิทยาการ สาขาวิชาพันธุวิศวกรรม บัณฑิตวิทยาลัย มหาวิทยาลัยเกษตรศาสตร์ ภาควิชาพันธุศาสตร์ คณะวิทยาศาสตร์ มหาวิทยาลัยเกษตรศาสตร์ ห้องปฏิบัติการเทคโนโลยีชีวภาพ สถาบันวิจัยจุฬาภรณ์ โทร 02-5625555 ต่อ 4203 อีเมล์ fscipns@ku.ac.th |